

본 게시글은 학부 '세포생물학' 강의와 강의 교재(The Cell : A Molecular Approach, Geoffrey M. Cooper)를 토대로 이해한 바를 정리하였습니다.
Contents
7.1 DNA Replication
- Compare the roles of DNA polymerases in E.coli with those in mammalian cells
- Contrast the mechanisms of synthesis of the leading and lagging strands of DNA
- Identify the proteins found at replication forks of bacteria and mammalian cells
- Describe the mechanisms that ensure accurate DNA replication
- Compare origins of replication in bacteria and mammalian cells
- Summarize the action of telomerase
7.2 DNA Repair
- Compare and contrast direct repair of DNA damage with the different types of excision repair
- Explain why defects in DNA repair lead to cancer
- Describe translesion DNA synthesis
- Summarize the mechanisms cells use to repair double-strand breaks
7.3 DNA Rearrangements and Gene Amplification
- Describe the rearrangements in immunoglobulin heavy and light chains
- Explain how nonhomologous end joining of double-strand breaks and vytosine deamination contribute to immunoglobulin diversity
- Explain why DNA amplification increases gene expression
7.1 DNA Replication
DNA Polymerases

진핵세포의 DNA polymerase
- α, δ ,ε : 핵 DNA 복제에 관여
- γ : 미토콘드리아에 위치하며, 미토콘드리아 DNA 복제를 담당
- 모든 DNA polymerase는 5'에서 3' 방향으로만 DNA 합성이 가능하다.
- DNA 합성을 시작하려면 반드시 Template에 수소결합된 Preformed primer가 필요하다.
The replication fork

복제 분기점(Replication fork)
- DNA 복제 중 활성 DNA 합성이 이루어지는 영역이다.
- 부모 가닥(Parental stands)이 분리되어 새로운 딸가닥(Daughter strands) 합성을 위한 Template으로 사용된다.
합성 가닥의 종류

- Leading strand : 선도가닥, 지속적으로 합성되는 가닥
- Lagging strand : 지연가닥, 역방향으로 불연속적으로 합성되는 짧은 DNA 조각(Okazaki fragments, 오카자키 절편)으로 구성
RNA primer

- Primase가 3-10 뉴클레오타이드 길이의 짧은 RNA fragment를 합성하여 복제를 시작한다.

- RNA primer의 제거방식
- 박테리아 : Polymerase I에 의해 제거된다.
- 진핵세포 : RNase H와 추가 단백질이 협력하여 제거된다.


- RFC(Clamp-loading protein) : Polymerase를 로딩하는 단백질
- PCNA(Sliding-clamp protein) : Polymerase가 primer에 결합하도록 돕는 단백질

Helicase
- ATP 가수분해를 통해 부모 DNA 가닥을 복제 분기접 앞에서 풀어준다.
Single-stranded DNA-binding proteins
- RPA : 풀어진 template DNA를 안정화시킨다.
Topoisomerase

- DNA 복제 중 발생하는 Supercoiling(초나선)을 해소한다.
- DNA 가닥을 가역적으로 절단하고 재결합시킨다.
- Type I : DNA 한 가닥을 절단
- Type II : DNA 두 가닥을 동시에 절단
The fidelity of replication

복제의 정확성
- DNA polymerase의 선택적 매커니즘
- DNA polymerase는 정확한 염기쌍(Conformation)에 맞게 적응하여 잘못된 염기(Mismatched base)가 삽입되는 것을 방지한다.
- DNA polymerase의 교정(Proofreading)
- 진핵 세포의 폴리머레이스 δ와 ε, 그리고 E.coli polymerase III는 3'에서 5' 방향으로 DNA를 가수분해하는 Exonuclease(외가닥가수분해) 활성을 통해 잘못된 염기를 제거하고 복제 정확성을 높인다.
Origins and the initiation of replication


복제 원점(Origins of Replication)
- 복제 개시 단백질이 결합하는 DNA 서열
- 복제는 여러 시작점에서 시작하며, 염색체를 따라 양방향으로 진행된다.

- 진핵생물 염색체의 복제 원점은 Yeast(효모)에서 처음 연구되었다.

ARSs(Autonomously Replicating Sequences)
- 다양한 ARS에서 공통적으로 발견되는 11 bp의 핵심 서열(Core sequence)를 포함하여 기능적 ARS 요소는 약 100 bp 길이를 가진다.
ORC(Origin Recognition Complex)
- 6개의 단위로 구성된 단백질 복합체로 ARS의 ACS(Autonomously Replicating Sequence)에 결합하여 복제를 시작한다.
- MCM DNA helicase와 같은 복제 단백질을 모이게 한다.
Telomeres and telomerase : Maintaining the ends of chromosomes


Telomeres
- 진핵세포의 선형 염색체의 말단 서열 복제를 담당한다.
- 단순 반복 서열로 구성된 tandem repeats(탠덤 반복)이 존재
Telomerase
- DNA template 없이 텔로미어를 합성한다.
- RNA template를 이용하여 DNA를 합성하는 역전사효소(reverse transcriptase) 역할을 수행한다.
- 자체 RNA template를 포함하며, telomere 반복 서열과 상보적이다.
7.2 DNA Repair
DNA damage


DNA는 다양한 화학적, 물리적 요인에 의해 손상될 수 있으며, 세포는 이를 복구하는 다양한 메커니즘을 보유하고 있다.
Direct reversal of DNA damage

UV에 의한 피리미딘 이량체(Pyrimidine Dimers)
- UV로 유도된 피리미딘 이량체를 직접 반응 역전으로 복구한다.
Photoreactivation(광재활성화)
- 가시광선에서 에너지를 사용하여 Cyclobutane ring 구조를 파괴하여 복구한다.
Excision repair(절제 수선)
- 다양한 화학적 손상을 복구하는 일반적 메커니즘
- 손상된 DNA르르 인식하여 제거하고, 결과적으로 남은 공백을 새로운 DNA 합성으로 채운다.
- 복구 기전으로는 크게 아래와 같다.
- 염기 절제 수선(Base-Excision Repair)
- 뉴클레오타이드 절제 수선(Nucleotide-Excision Repair)
- 미스매치 수선(Mismatch Repair)
Base-Excision Repair(염기 절제 수선)

- e.g. DNA에 포함된 Uracil(유라실) 복구 기작
- DNA glycosylase - 손상된 염기(유라실)와 디옥시리보스 사이의 결합 절단
- AP endonuclease - AP 자리(손상된 염기가 제거된 자리) 인접부 절단
- Deoxyribose phospho diesterase - 남은 디옥시리보오스를 제거
- DNA polymerase와 ligase - 단일 염기 공백을 채우고 연결
Nucleotide-Excision Repair(뉴클레오타이드 절제 수선)

- 손상된 염기를 포함한 oligonucleotide 전체를 제거한다.
- XPA ~ XPG로 명명된 7개의 수선 유전자가 관여한다.
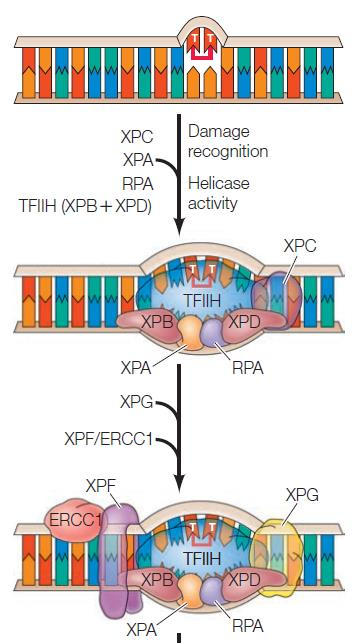

Mismatch Repair(미스매치 수선)


- 새로 복제된 DNA에서 발생한 잘못된 염기쌍(Mismatched base)을 제거한다.
- 복제 후 DNA가 메틸화되기 전 발생한다.
- MSH와 MLH : 잘못된 염기와 결합하여 절제를 유도한다.
Translesion DNA synthesis

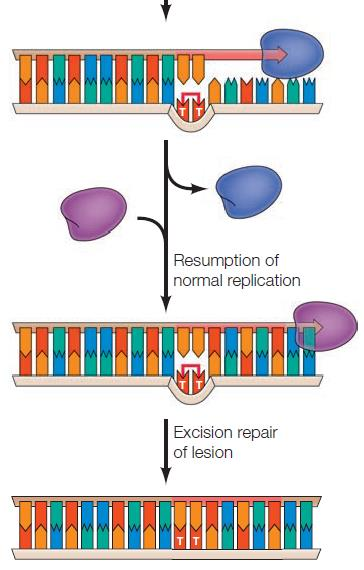
- 복제 분기점에서 손상된 DNA를 우회하여 복제하는 매커니즘
- 이후 복제가 완료된 후 손상을 수정한다.
- DNA polymerase V 가 관여한다.
Repair of double-strand breaks

Double-strand breaks(이중 가닥 절단)
- DNA 분자가 두 가닥 모두 절단되어 가장 위험한 형태의 손상
수선 기작
- Recombinational Repair(재조합 복구) - 끊어진 가닥을 재결합
- 오류가 발생할 가능성이 높다.

- Homologous Recombination(상동 재조합) - 손상되지 않은 염색체의 DNA 서열을 이용해 수선
7.3 DNA Rearrangements and Gene Amplification
Antibody genes

항체 구조
- 항체는 동일한 무거운 사슬(Heavy chain)과 가벼운 사슬(Light chain)로 이루어진다.
항체와 림프구
- B 림프구 : 항체(면역 글로불린)를 분비하여 용해성 항원과 반응한다.
- T 림프구 : T 세포 수용체(T cell receptors)를 발현하며, 다른 세포 표면의 항원과 반응한다.

각 폴리펩타이드 사슬에는 다음의 영역이 포함된다.
- V(Variable) 영역 : 폴리펩타이드 가변 영역의 N-말단 95-96개 아미노산을 암호화
- J(Joining) 영역 : 가변 영역의 C-말단 12-14개 아미노산을 암호화
- C(Constant) 영역 : 폴리펩타이드의 일정 영역을 암호화
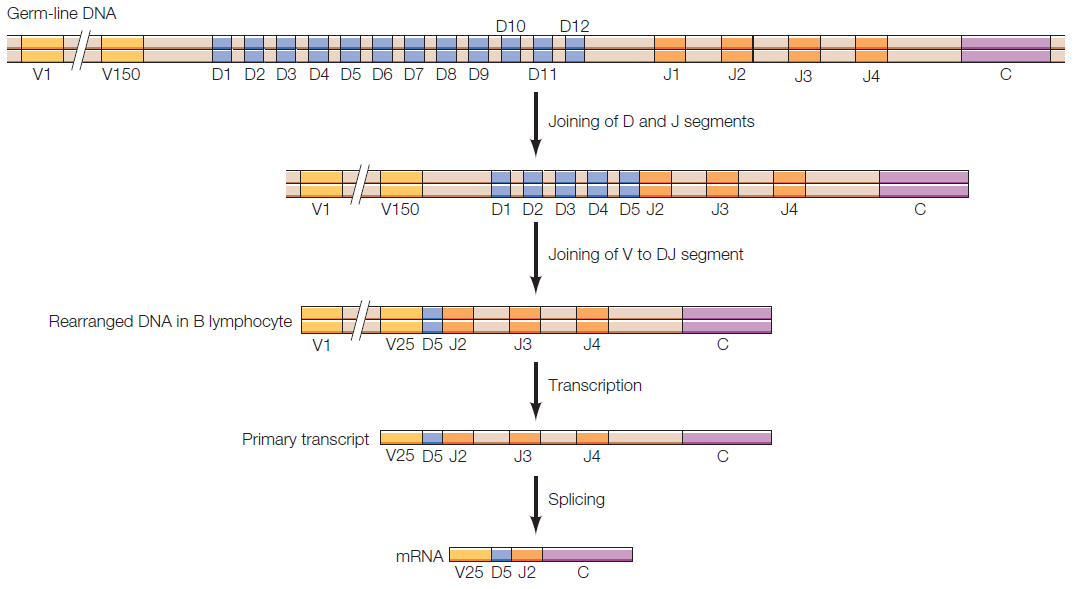
- D(Diversity) 영역(Heavy chain only) : V와 J 사이의 아미노산을 암호화

T 세포 수용체
- α 사슬 : V와 J 재조합으로 생성
- β 사슬 : V, D, J 재조합으로 생성
- 이는 항체 유전자 형성과 유사한 방식으로 유전자 재조합을 통해 생성된다.

- RAG protein : 각 유전자 segment의 coding sequence에 인접한 RS(재조합 신호) 서열을 인식한다.
- RS와 coding sequence 사이에 이중 가닥 절단을 도입하여 DNA 재배열을 시작한다.

Class switch Recombination(클래스 스위치 재조합)
재조합된 가변 영역을 새로운 하부 Constant Region과 재배열하고, 중간 DNA를 삭제한다.
체성 과돌연변이)Somatic Hypermutation)
재조합된 가변 영역(무거운 사슬과 가벼운 사슬)에 다수의 돌연변이를 유도하여 면역글로불린의 다양성을 증가시킨다.

- AID(Activation-Induced Deaminase) : B 림프구에서만 발현되며, 클래스 스위치 재조합과 체성 과돌연변이에 필요하다.
Gene amplification
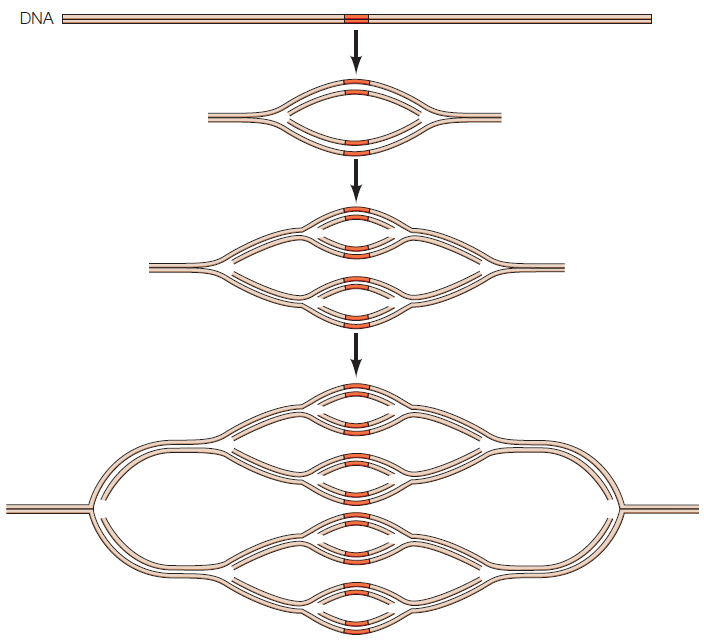
- 특정 DNA 영역의 반복적 복제로 인해 해당 영역의 복사본 수가 증가하는 현상
- 유전자 증폭은 유전자 발현을 증가시킨다.
- 정상적으로 발달 과정에서 발생하거나, 암세포에서 비정상적으로 나타날 수 있다.
'BIOLOGY > Cell Biology' 카테고리의 다른 글
| Chapter 9. Transcriptional Regulation and Epigenetics (1) | 2024.12.09 |
|---|---|
| Chapter 8. RNA Synthesis and Processing (0) | 2024.12.08 |
| Chapter 6. Genes and Genomes (0) | 2024.12.07 |
| Chapter 5. Genomics, Proteomics, and Systems Biology (1) | 2024.12.06 |
| CNU Conference of Advanced Technology(CCAT) (0) | 2024.12.05 |

나의 성장 드라마
포스팅이 좋았다면 "좋아요❤️" 또는 "구독👍🏻" 해주세요!