

내가 원하는 건 돈 복사지
유전자 복사가 아니란 말이다!
Table of contents
1. DNA Transactions during Replication
- Semiconservative replication
- Replication fork
- Semidiscontinuous replication
2. The Chemistry of DNA Polymerase
- DNA polymerase I
- The role of metal ion
3. Mechanics of the DNA Replication Fork
- The protein complex in replication fork
4. Initiation of DNA Replication
- Assembly of replication fork
5. Termination of DNA Replication
- Telomere
유전자는 레서피라 했다. 내 자신을 이렇게 만들기 위한 레서피. 그 많은 세포끼리 레서피 한 권으로 돌려보기는 턱없이 모자랄 터. 그래서 세포는 최고급 복사기를 하나 장만했다. 근데 그 복사기가 너무 쓰기가 복잡하더라. 너무 성능이 좋아 모든 걸 그대로 베껴버리더라. 원하는 부분은 발췌도 해주기도 한다. 그래서 우리는 이 복사기의 사용법을 알아보려 한다.
그렇게 최첨단이면 내 통장의 돈도 좀 복사 안되나..?
Semiconservative model by Watson & Crick

이중가닥 DNA가 벌어져 한 가닥을 주형으로 나머지 새로운 가닥이 합성된다.
DNA 모델이 완성된 후 복제 방법에 대해 다양한 의견들이 제시되었다.


이를 증명하기 위해 안정성 동위원소가 사용되었는데 부모 DNA에는 15N DNA로 표지하였고, 복제된 DNA에는 14N DNA로 표지되도록 하였다. 그림에서 보여주듯 14N은 15N보다 상대적으로 가볍기 때문에 원심분리(Centrifuge)를 진행하면 가벼운 만큼 15N보다 위에 위치하게 된다.

하지만 실험을 통해 관찰된 결과 Semiconservative replication 모델과 일치하는 모습을 보여주었다.
Question)
Synthesis initiated at one site or at multiple sites on the chromosome ?
The sites are defined or random?
-> 하지만 이 질문에 대해서, 미래에서 온 우리는 이미 답을 알고 있다.
Replication Origin(복제 원점)에 의해 결정되어 있음을 우리는 안다.

Origin of replication : 복제 원점(OriC). A=T 염기쌍이 풍부하다. 여기에 DnaA 단백질이 결합한다.
Replication fork : 복제 분기점. 복제 중인 DNA에서 관찰되는 Y자 모양의 분기점.
Theta (θ) form replication : θ 모양의 형태가 관찰된다. 원형 플라스미드 DNA가 복제될 때 중간 단계에서 θ 구조가 형성한다. θ형 복제에서는 두 DNA 가닥이 동시에 복제되고 replication fork이 양방향으로 이동. 양쪽 가닥 모두에서 새로운 DNA가 합성한다.

여기서 포인트는 양방향으로 진행(Bidirectional)되고 여러 개의 출발점에서 복제가 시작된다는 것(Multiple sites)이다.
원핵생물에서는 OriC가 하나인 점과는 다르다.

- 오카자키 절편이 생기는 이유 ⇒ 5’ → 3’으로 polymerase가 진행하기 때문
- 오카자키 절편은 진핵세포가 더 짧다. ⇒ 합성 속도가 느리다.
Semidiscontinuous replication으로 인해 Leading strand,Lagging Strand가 생긴다. Lagging strand의 범주에 okazaki fragment가 속해 있다.
Lagging strand는 3’ → 5’으로 합성되어야 하지만 그럴 수 없기 때문에 불연속적으로 합성된다.

The Chemistry of DNA Polymerase

dNTP에서 PPi가 떨어져나오면서 방출되는 에너지를 이용해서 염기가 연결된다.


결국 Nick을 제거하려면 Polymerase III가 RNA primer까지 제거한 뒤, ligase가 그 nick까지 메꿔주어야 한다.
Pol I의 Structure과 form


손가락 모양처럼 생겼다. 하지만 행동또한 손과 흡사하다. Open form의 형태에서 검지와 중지처럼 보이는 사이에 붙일 뉴클레오타이드를 하나 가지고 있다가 ATP를 소모하며 closed form이 형성되면서 DNA에 염기를 연결하게 된다.
- dNTP가 Pol I의 활성 부위에 접근, 이때 Pol I는 open form 상태로 활성 부위가 노출되어 있다.
- dNTP가 주형 DNA 가닥과 상보적으로 결합, 그리고 올바르게 염기쌍이 dNTP와 주형 DNA 가닥 사이에 형성되면 Pol I 구조가 변화한다.
- 올바르게 염기쌍이 형성되면 Pol I은 close form으로 변하고 이는 효소의 finger domain이 회전하여 dNTP와 주형 DNA 가닥을 둘러싸게 한다. 이로 인해 활성 부위가 더 정확히 맞물리게 되어 효소의 촉매 효율이 증가한다.
- Close form 상태에서 Pol I은 dNTP의 인산 결합을 형성하고 새로운 nucleotide를 DNA 사슬에 추가하고 반응이 완료되면 효소는 다시 open form으로 돌아가 다음 dNTP를 받아들일 준비를 한다.

- Induced Fit Model : 효소와 기질이 결합할 때, 효소의 활성 부위가 구조적으로 기질에 맞게 되는 모델(유도 적합 모델)
- Pol I의 활성 부위(Active-site)
- 올바른 염기쌍 - DNA 주형 가닥과 상보적인 뉴클레오타이드와 결합할 때, Pol I의 활성 부위는 close form 으로 변형되어 안정적인 결합 형성
- 잘못된 염기쌍 - 상보적이지 않은 뉴클레오타이드가 결합할 때, Pol I 활성 부위는 구조적으로 불안정해지며, 결합이 느슨해진다.
잘못된 염기쌍이 형성되면, Pol I는 구조적으로 불안정해지기 때문에 Active site가 닫히지 않는다. 이는 잘못된 뉴클레오타이드가 방출되거나 제거되는 과정이 촉진된다.
금속 이온(Mg²⁺)의 DNA 폴리메라아제 촉매 작용에서의 역할

(a) DNA synthesis
- 활성화와 안정화
- 인산기 안정화 : 두 개의 Mg²⁺이온이 dNTP의 인산기(그 중 알파 인산기)를 안정화 시켜 반응 준비를 돕는다.
- 3’-OH 그룹 활성화 : 하나의 Mg²⁺ 이온은 성장하는 DNA 가닥의 3’- OH 그룹을 활성화시켜 인산다이에스터 결합 형성을 촉진한다. 이는 3’-OH 그룹의 deprotonation(탈양성자화)를 통해 이루어진다.
- 촉매 활성화
- 친핵성 공격 촉진 : 활성화된 3’-OH 그룹은 인접한 dNTP의 알파 인산기에 친핵성 공격을 하여 인산 다이에스터 결합을 형성한다.
- 피로인산 방출 : 이 반응에서 두 번째 Mg²⁺ 이온은 생성된 피로인산의 안정화를 돕고, 반응 후 방출을 촉진한다.
(b) 3’ → 5’ exonuclease
- 오류 검출 및 수정
- 잘못된 염기 제거 : 잘못 삽입된 뉴클레오타이드는 polymerase의 active site에서 인식되어 exonuclease 활성 부위로 이동한다.
- Mg²⁺ 이온의 역할
- 수산화 이온 활성화 : 두 개의 Mg²⁺ 이온이 존재, 이들은 물 분자를 탈양성자화하여 활성화된 OH-를 형성한다.
- 핵산 가수분해 촉진 : 활성화된 OH-은 잘못된 뉴클레오타이드의 인산디에스터 결합을 가수분해하여 잘못된 뉴클레오타이드를 제거한다.
- Mg²⁺이온 2. 수산화 이온을 조정하여 인산다이에스터 결합 가수분해

α Subunit은 Pol lll holoenzyme(복합체)에서 매우 핵심적인 역할 수행한다.
- DNA 합성
- 복제 진행
- 연속적 합성 : α Subunit은 선도가닥과 지연 가닥 모두에서 DNA 합성 촉진
- 오카자키 절편 합성

The E. coli Pol III holoenzyme
-> 3 Pol III core, 3 ring-shaped b sliding clamps, 1 clamp loader
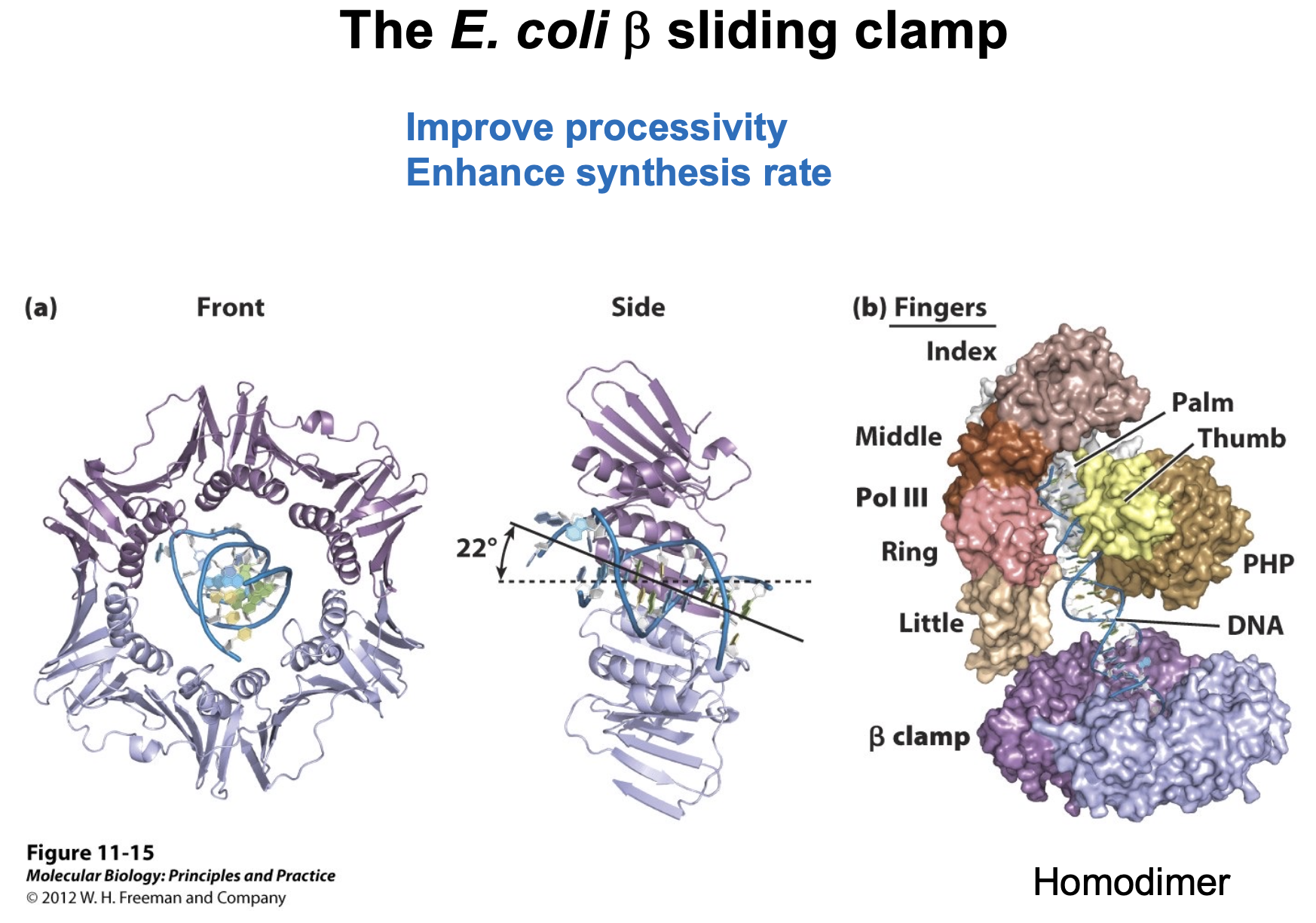


DNA pol lll holoenzyme은 다중 단백질 복합체.
- Pol lll Core
- α, ε, θ 서브유닛으로 구성
- α subunit : 주된 DNA 합성 기능 담당
- ε subunit : 3’ → 5’ 방향의 proof-reading exonuclease 수행. 잘못 삽입된 뉴클레오타이드 제거하여 합성 정확성 증가
- θ subunit : ε subunit의 안정성과 기능을 강화하는 보조 역할
- α, ε, θ 서브유닛으로 구성
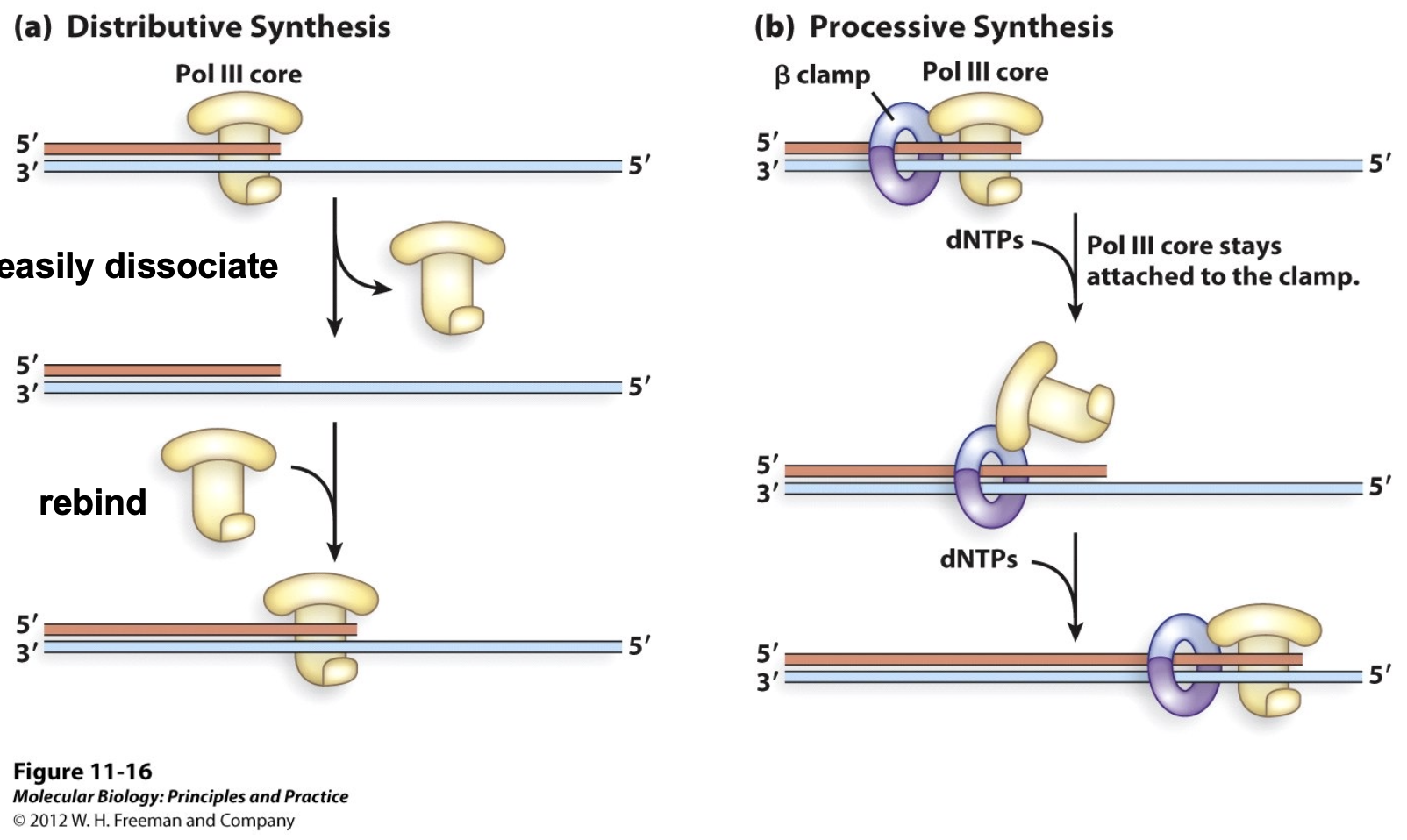
- β Sliding Clamp
- 두 개의 subunit이 링 모양으로 결합. Pol lll core가 DNA에 더 강하게 결합할 수 있도록 한다. DNA가 합성하는 동안 Polymerase가 DNA 주형 가닥에 떨어지지 않고 지속적으로 합성할 수 있게한다.
- β Sliding Clamp는 Clamp loader에 의해 DNA에 결합되며, 결합 후에는 polymerase와 함께 DNA를 따라 이동한다.
- Clamp loader
- ATP를 이용하여 β Clamp를 열고, Clamp가 DNA를 둘러싸게 한다. Clamp loader는 β Clamp를 DNA에 결합시키고, 결합이 완료되면 해리된다.
해리된 β Sliding Clamp는 다시 clamp loader와 결합하여 다음 오카자키 절편 또는 새로운 복제 시작점에 loading 된다.
Hypothetical model of the eukaryotic replication fork
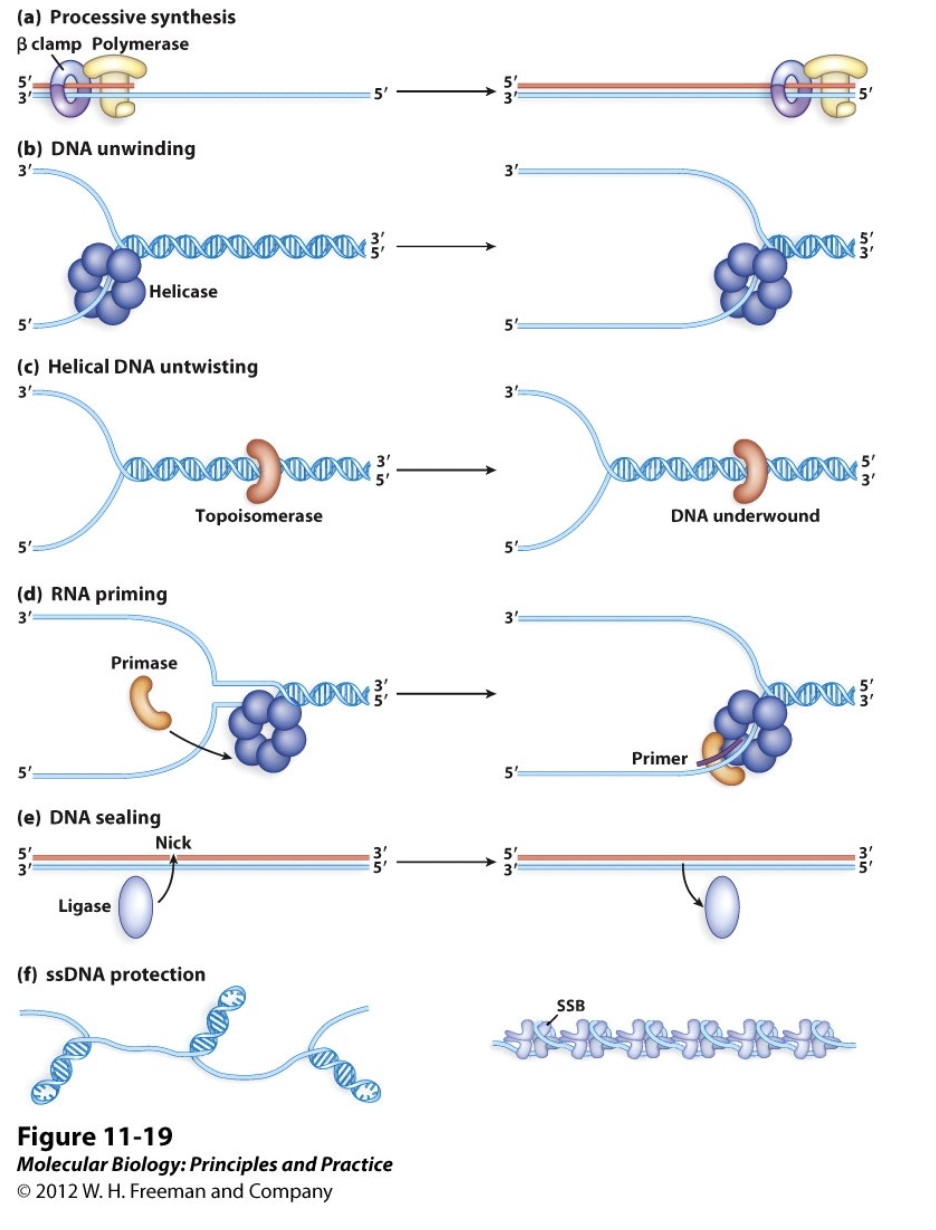
원핵생물과 진핵생물에 각각 하는 일은 비슷하지만 지칭하는 용어가 다르다. 분석해보면 엄연히 다른 물질이니까.
The replication fork is more complex in eukaryotes than in bacteria
Many eukaryotic replication proteins have counterparts in bacteria
MCM helicase = DnaB helicase
activity direction (3’ -> 5’, leading strand)
Pol a = primase
PCNA = b sliding clamp
RFC (replication factor C) = clamp loader RPB (replication protein A) = SSB
'BIOLOGY > Molecular Biology' 카테고리의 다른 글
| Mutation - 추후 설명 보충 예정 (0) | 2024.06.18 |
|---|---|
| DNA Replication season 2 (0) | 2024.06.18 |
| Chemical Basis of Information Molecules - 추후보충 (0) | 2024.04.30 |
| DNA: The repository of Biological Information (2) | 2024.03.26 |
| Evolution, science and molecular biology (0) | 2024.03.24 |

나의 성장 드라마
포스팅이 좋았다면 "좋아요❤️" 또는 "구독👍🏻" 해주세요!



